+検索条件
-Structure paper
| タイトル | Neutralization, effector function and immune imprinting of Omicron variants. |
|---|---|
| ジャーナル・号・ページ | Nature, Vol. 621, Issue 7979, Page 592-601, Year 2023 |
| 掲載日 | 2023年8月30日 |
 著者 著者 | Amin Addetia / Luca Piccoli / James Brett Case / Young-Jun Park / Martina Beltramello / Barbara Guarino / Ha Dang / Guilherme Dias de Melo / Dora Pinto / Kaitlin Sprouse / Suzanne M Scheaffer / Jessica Bassi / Chiara Silacci-Fregni / Francesco Muoio / Marco Dini / Lucia Vincenzetti / Rima Acosta / Daisy Johnson / Sambhavi Subramanian / Christian Saliba / Martina Giurdanella / Gloria Lombardo / Giada Leoni / Katja Culap / Carley McAlister / Anushka Rajesh / Exequiel Dellota / Jiayi Zhou / Nisar Farhat / Dana Bohan / Julia Noack / Alex Chen / Florian A Lempp / Joel Quispe / Lauriane Kergoat / Florence Larrous / Elisabetta Cameroni / Bradley Whitener / Olivier Giannini / Pietro Cippà / Alessandro Ceschi / Paolo Ferrari / Alessandra Franzetti-Pellanda / Maira Biggiogero / Christian Garzoni / Stephanie Zappi / Luca Bernasconi / Min Jeong Kim / Laura E Rosen / Gretja Schnell / Nadine Czudnochowski / Fabio Benigni / Nicholas Franko / Jennifer K Logue / Courtney Yoshiyama / Cameron Stewart / Helen Chu / Hervé Bourhy / Michael A Schmid / Lisa A Purcell / Gyorgy Snell / Antonio Lanzavecchia / Michael S Diamond / Davide Corti / David Veesler /     |
| PubMed 要旨 | Currently circulating SARS-CoV-2 variants have acquired convergent mutations at hot spots in the receptor-binding domain (RBD) of the spike protein. The effects of these mutations on viral infection ...Currently circulating SARS-CoV-2 variants have acquired convergent mutations at hot spots in the receptor-binding domain (RBD) of the spike protein. The effects of these mutations on viral infection and transmission and the efficacy of vaccines and therapies remains poorly understood. Here we demonstrate that recently emerged BQ.1.1 and XBB.1.5 variants bind host ACE2 with high affinity and promote membrane fusion more efficiently than earlier Omicron variants. Structures of the BQ.1.1, XBB.1 and BN.1 RBDs bound to the fragment antigen-binding region of the S309 antibody (the parent antibody for sotrovimab) and human ACE2 explain the preservation of antibody binding through conformational selection, altered ACE2 recognition and immune evasion. We show that sotrovimab binds avidly to all Omicron variants, promotes Fc-dependent effector functions and protects mice challenged with BQ.1.1 and hamsters challenged with XBB.1.5. Vaccine-elicited human plasma antibodies cross-react with and trigger effector functions against current Omicron variants, despite a reduced neutralizing activity, suggesting a mechanism of protection against disease, exemplified by S309. Cross-reactive RBD-directed human memory B cells remained dominant even after two exposures to Omicron spikes, underscoring the role of persistent immune imprinting. |
 リンク リンク |  Nature / Nature /  PubMed:37648855 / PubMed:37648855 /  PubMed Central PubMed Central |
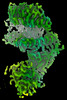
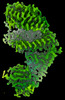
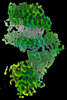
| 手法 | EM (単粒子) |
| 解像度 | 3.0 - 3.2 Å |
| 構造データ | EMDB-29530, PDB-8fxb: EMDB-29531, PDB-8fxc: EMDB-40240, PDB-8s9g: |
| 化合物 | 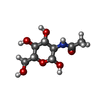 ChemComp-NAG:  ChemComp-ZN: |
| 由来 |
|
 キーワード キーワード |  VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) / VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) /  SARS-CoV-2 (SARSコロナウイルス2) / SARS-CoV-2 (SARSコロナウイルス2) /  COVID-19 (新型コロナウイルス感染症 (2019年)) / XBB.1 / COVID-19 (新型コロナウイルス感染症 (2019年)) / XBB.1 /  Spike glycoprotein (スパイクタンパク質) / Spike glycoprotein (スパイクタンパク質) /  Neutralizing antibodies (中和抗体) / Neutralizing antibodies (中和抗体) /  Structural Genomics (構造ゲノミクス) / Seattle Structural Genomics Center for Infectious Disease / SSGCID / Inhibitor / Structural Genomics (構造ゲノミクス) / Seattle Structural Genomics Center for Infectious Disease / SSGCID / Inhibitor /  ACE2 (アンジオテンシン変換酵素2) / ACE2 (アンジオテンシン変換酵素2) /  VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) / BQ.1.1 / VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) / BQ.1.1 /  VIRAL PROTEIN (ウイルスタンパク質) / BN.1 VIRAL PROTEIN (ウイルスタンパク質) / BN.1 |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について