+検索条件
-Structure paper
| タイトル | PtuA and PtuB assemble into an inflammasome-like oligomer for anti-phage defense. |
|---|---|
| ジャーナル・号・ページ | Nat Struct Mol Biol, Vol. 31, Issue 3, Page 413-423, Year 2024 |
| 掲載日 | 2024年1月4日 |
 著者 著者 | Yuanyuan Li / Zhangfei Shen / Mengyuan Zhang / Xiao-Yuan Yang / Sean P Cleary / Jiale Xie / Ila A Marathe / Marius Kostelic / Jacelyn Greenwald / Anthony D Rish / Vicki H Wysocki / Chong Chen / Qiang Chen / Tian-Min Fu / Yamei Yu /   |
| PubMed 要旨 | Escherichia coli Septu system, an anti-phage defense system, comprises two components: PtuA and PtuB. PtuA contains an ATPase domain, while PtuB is predicted to function as a nuclease. Here we show ...Escherichia coli Septu system, an anti-phage defense system, comprises two components: PtuA and PtuB. PtuA contains an ATPase domain, while PtuB is predicted to function as a nuclease. Here we show that PtuA and PtuB form a stable complex with a 6:2 stoichiometry. Cryo-electron microscopy structure of PtuAB reveals a distinctive horseshoe-like configuration. PtuA adopts a hexameric arrangement, organized as an asymmetric trimer of dimers, contrasting the ring-like structure by other ATPases. Notably, the three pairs of PtuA dimers assume distinct conformations and fulfill unique roles in recruiting PtuB. Our functional assays have further illuminated the importance of the oligomeric assembly of PtuAB in anti-phage defense. Moreover, we have uncovered that ATP molecules can directly bind to PtuA and inhibit the activities of PtuAB. Together, the assembly and function of the Septu system shed light on understanding other ATPase-containing systems in bacterial immunity. |
 リンク リンク |  Nat Struct Mol Biol / Nat Struct Mol Biol /  PubMed:38177683 PubMed:38177683 |
| 手法 | EM (単粒子) |
| 解像度 | 2.6 - 2.93 Å |
| 構造データ | EMDB-28045, PDB-8ee4: EMDB-28048, PDB-8ee7: EMDB-28049, PDB-8eea: EMDB-40779, PDB-8sux: |
| 化合物 | 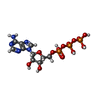 ChemComp-ATP: |
| 由来 |
|
 キーワード キーワード |  IMMUNE SYSTEM (免疫系) / PtuA / PtuB Septu / anti-phage / Septu system IMMUNE SYSTEM (免疫系) / PtuA / PtuB Septu / anti-phage / Septu system |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について












