-検索条件
-検索結果
検索 (著者・登録者: luo & t)の結果633件中、1から50件目までを表示しています

EMDB-37663: 
CryoEM structure of ZIKV rsNS1

EMDB-37670: 
ZIKV rsNS1 in complex with Fab AA12 and anti-fab nanobody

EMDB-37673: 
ZIKV rsNS1 in complex with Fab GB5 and anti-fab nanobody

EMDB-37676: 
CryoEM structure of ZIKV rsNS1 filament

EMDB-37678: 
ZIKV rsNS1 in complex with Fab EB9 and anti-fab nanobody

EMDB-38999: 
Focused refinement map of BRR2 region of the human minor pre-B complex

EMDB-37104: 
96-nm axonemal repeat with RS1/2/3

EMDB-37111: 
48-nm repeat DMT

EMDB-37114: 
Radial Spoke 1 (RS1)
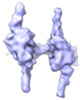
EMDB-37116: 
RS1 refined with head mask

EMDB-37117: 
Radial Spoke 2 (RS2)
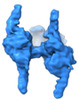
EMDB-37118: 
Radial Spoke 2 (RS2) head

EMDB-37119: 
Radial Spoke 3

EMDB-37120: 
Radial Spoke 3 head

EMDB-39005: 
Focused refinement map of U5 snRNP of the human minor pre-B complex

EMDB-39006: 
Focused refinement map of U5 Sm ring region of the human minor pre-B complex

EMDB-39007: 
Focused refinement map for U4atac/U6atac region of the human minor pre-B complex

EMDB-36800: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state

EMDB-36801: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrA octamer

EMDB-36802: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrB dimer

EMDB-36803: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of MgCl2

EMDB-36804: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA

EMDB-38477: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, vertical C2 symmetry axis

EMDB-38478: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, C1 symmetry
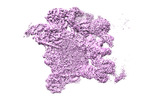
EMDB-38993: 
Cryo-EM Structure of the human minor pre-B complex (pre-precatalytic spliceosome) U11 and tri-snRNP part

EMDB-39000: 
focused refinement map for the U11 snRNP in the human minor pre-B complex

PDB-8y6o: 
Cryo-EM Structure of the human minor pre-B complex (pre-precatalytic spliceosome) U11 and tri-snRNP part

EMDB-37690: 
Structure of the wild-type Arabidopsis ABCB19 in the apo state

EMDB-37692: 
Structure of the wild-type Arabidopsis ABCB19 in the brassinolide-bound state

EMDB-37694: 
Structure of the wild-type Arabidopsis ABCB19 in the brassinolide and AMP-PNP bound state

EMDB-37705: 
Structure of the Arabidopsis E529Q/E1174Q ABCB19 in the ATP bound state

PDB-8woi: 
Structure of the wild-type Arabidopsis ABCB19 in the apo state

PDB-8wom: 
Structure of the wild-type Arabidopsis ABCB19 in the brassinolide-bound state

PDB-8woo: 
Structure of the wild-type Arabidopsis ABCB19 in the brassinolide and AMP-PNP bound state

PDB-8wp0: 
Structure of the Arabidopsis E529Q/E1174Q ABCB19 in the ATP bound state
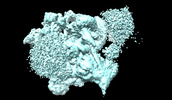
EMDB-39013: 
Cryo-EM Structure of the human minor pre-B complex (pre-precatalytic spliceosome) U12 snRNP part

PDB-8y7e: 
Cryo-EM Structure of the human minor pre-B complex (pre-precatalytic spliceosome) U12 snRNP part

EMDB-37390: 
Cryo-EM structure of peptide free and Gs-coupled GIPR

EMDB-37504: 
Cryo-EM structures of peptide free and Gs-coupled GLP-1R
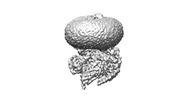
EMDB-37505: 
Cryo-EM structures of peptide free and Gs-coupled GCGR

PDB-8wa3: 
Cryo-EM structure of peptide free and Gs-coupled GIPR

PDB-8wg7: 
Cryo-EM structures of peptide free and Gs-coupled GLP-1R

PDB-8wg8: 
Cryo-EM structures of peptide free and Gs-coupled GCGR
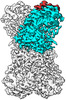
EMDB-34200: 
Complex Structure of Arginine Kinase McsB and McsA from Staphylococcus aureus

PDB-8gqd: 
Complex Structure of Arginine Kinase McsB and McsA from Staphylococcus aureus
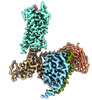
EMDB-36144: 
Senktide bound to active human neurokinin 3 receptor in complex with Gq

PDB-8jbf: 
Senktide bound to active human neurokinin 3 receptor in complex with Gq

EMDB-28809: 
Yeast ATP synthase map in conformation-1 at pH 6

EMDB-28835: 
Yeast ATP synthase map in conformation-2, at pH 6

PDB-8f29: 
Yeast ATP synthase in conformation-1 at pH 6
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
