-検索条件
-検索結果
検索 (著者・登録者: sobolevsky & ai)の結果131件中、1から50件目までを表示しています

EMDB-44124: 
Structure of concanavalin A (ConA) dimer from the open-state structure of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to one ConA dimer. Type II interface between GluK2 ligand-binding domain and ConA

EMDB-44125: 
Structure of concanavalin A (ConA) dimer from the open-state structure of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to two ConA dimers. Type I interface between GluK2 ligand-binding domain and ConA

EMDB-44128: 
Ligand-binding and transmembrane domains of kainate receptor GluK2 in the open state, a complex with agonist glutamate and positive allosteric modulator BPAM344

EMDB-44129: 
Open state of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to two concanavalin A dimers. Composite map.

EMDB-44130: 
Open state of kainate receptor GluK2 in complex with agonist glutamate and positive allosteric modulator BPAM344 bound to one concanavalin A dimer. Composite map.

EMDB-44131: 
Kainate receptor GluK2 in complex with agonist glutamate with pseudo 4-fold symmetrical ligand-binding domain layer

EMDB-44132: 
Kainate receptor GluK2 in complex with agonist glutamate with asymmetric ligand-binding domain layer
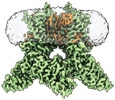
EMDB-42994: 
Apo-state cryo-EM structure of human TRPV3 in cNW30 nanodiscs
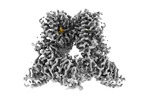
EMDB-42995: 
Open-state cryo-EM structure of human TRPV3 in presence of tetrahydrocannabivarin (THCV) in cNW30 nanodiscs

EMDB-42996: 
Inactivated-state cryo-EM structure of human TRPV3 in presence of tetrahydrocannabivarin (THCV) in cNW30 nanodiscs

EMDB-42997: 
Open-state cryo-EM structure of human TRPV3 in presence of 2-APB in cNW30 nanodiscs

EMDB-42998: 
Inactivated-state cryo-EM structure of human TRPV3 in presence of 2-APB in cNW30 nanodiscs

EMDB-43751: 
TRPM7 structure in complex with anticancer agent CCT128930 in closed state

EMDB-16052: 
Cryo-EM structure of stalled rabbit 80S ribosomes in complex with human CCR4-NOT and CNOT4

EMDB-40741: 
Structure of AMPA receptor GluA2 complex with auxiliary subunits TARP gamma-5 and cornichon-2 bound to competitive antagonist ZK and channel blocker spermidine (closed state)

EMDB-40742: 
Structure of LBD-TMD of AMPA receptor GluA2 in complex with auxiliary subunits TARP gamma-5 and cornichon-2 bound to competitive antagonist ZK and channel blocker spermidine (closed state)

EMDB-40743: 
Structure of LBD-TMD of AMPA receptor GluA2 in complex with auxiliary subunits TARP gamma-5 and cornichon-2 (apo state)

EMDB-40744: 
Structure of LBD-TMD of AMPA receptor GluA2 in complex with auxiliary subunit TARP gamma-5 (apo state)

EMDB-40745: 
Structure of AMPA receptor GluA2 complex with auxiliary subunits TARP gamma-5 and cornichon-2 bound to competitive antagonist ZK, channel blocker spermidine and antiepileptic drug perampanel (closed state)

EMDB-40746: 
Structure of AMPA receptor GluA2 complex with auxiliary subunits TARP gamma-5 and cornichon-2 bound to competitive antagonist ZK, channel blocker spermidine and antiepileptic drug perampanel (closed state)

EMDB-40747: 
Structure of AMPA receptor GluA2 complex with auxiliary subunit TARP gamma-5 bound to competitive antagonist ZK and antiepileptic drug perampanel (closed state)

EMDB-40748: 
Structure of LBD-TMD of AMPA receptor GluA2 in complex with auxiliary subunit TARP gamma-5 bound to competitive antagonist ZK and antiepileptic drug perampanel (closed state)

EMDB-40749: 
Structure of AMPA receptor GluA2 complex with auxiliary subunits TARP gamma-5 and cornichon-2 bound to glutamate and channel blocker spermidine (desensitized state)

EMDB-40750: 
Structure of LBD-TMD of AMPA receptor GluA2 in complex with auxiliary subunits TARP gamma-5 and cornichon-2 bound to glutamate and channel blocker spermidine (desensitized state)
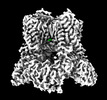
EMDB-40676: 
Human TRP channel TRPV6 in cNW30 nanodiscs inhibited by tetrahydrocannabivarin (THCV)

EMDB-40958: 
Cryo-EM structure of full-length human TRPV4 in apo state

EMDB-40959: 
Cryo-EM structure of human TRPV4 ankyrin repeat domain in complex with GTPase RhoA
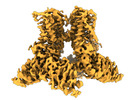
EMDB-40960: 
Open-state cryo-EM structure of full-length human TRPV4 in complex with agonist 4a-PDD

EMDB-40961: 
Closed-state cryo-EM structure of full-length human TRPV4 in the presence of 4a-PDD

EMDB-40962: 
Cryo-EM structure of full-length human TRPV4 in complex with antagonist HC-067047

EMDB-29343: 
Cryo-EM structure of human TRPV6 in complex with the natural phytoestrogen genistein

EMDB-29344: 
Cryo-EM structure of human TRPV6 in the open state

EMDB-40496: 
Cryo-EM structure of TRPM7 in MSP2N2 nanodisc in apo state

EMDB-40497: 
Cryo-EM structure of TRPM7 in GDN detergent in apo state

EMDB-40498: 
Cryo-EM structure of TRPM7 N1098Q mutant in GDN detergent in open state

EMDB-40499: 
Cryo-EM structure of TRPM7 in MSP2N2 nanodisc in complex with agonist naltriben in open state
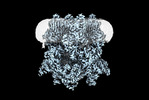
EMDB-40500: 
Cryo-EM structure of TRPM7 in MSP2N2 nanodisc in complex with agonist naltriben in closed state
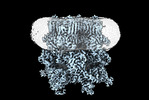
EMDB-40501: 
Cryo-EM structure of TRPM7 in GDN detergent in complex with inhibitor VER155008 in closed state

EMDB-40502: 
Cryo-EM structure of TRPM7 N1098Q mutant in GDN detergent in complex with inhibitor VER155008 in closed state

EMDB-40504: 
Cryo-EM structure of TRPM7 N1098Q mutant in GDN detergent in complex with inhibitor NS8593 in closed state

EMDB-40505: 
Cryo-EM structure of TRPM7 MHR1-3 domain

EMDB-29981: 
Cryo-EM structure of human TRPV1 in cNW11 nanodisc and soybean lipids

EMDB-29982: 
Cryo-EM structure of human TRPV1 in cNW11 nanodisc and POPC:POPE:POPG lipids

EMDB-29983: 
Cryo-EM structure of human TRPV1 in complex with the analgesic drug SB-366791

PDB-8gf8: 
Cryo-EM structure of human TRPV1 in cNW11 nanodisc and soybean lipids

PDB-8gf9: 
Cryo-EM structure of human TRPV1 in cNW11 nanodisc and POPC:POPE:POPG lipids

PDB-8gfa: 
Cryo-EM structure of human TRPV1 in complex with the analgesic drug SB-366791

EMDB-29515: 
Structure of kainate receptor GluK2 in complex with the positive allosteric modulator BPAM344

EMDB-29516: 
Structure of the amino-terminal domain of kainate receptor GluK2 in complex with the positive allosteric modulator BPAM344

EMDB-29517: 
Structure of the ligand-binding and transmembrane domains of kainate receptor GluK2 in complex with the positive allosteric modulator BPAM344
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
