-検索条件
-検索結果
検索 (著者・登録者: patel & h)の結果443件中、1から50件目までを表示しています

EMDB-29950: 
SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 002-S21B10

EMDB-29975: 
Overall map of SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 002-S21B10

EMDB-40007: 
Local map of SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 002-S21B10

EMDB-28809: 
Yeast ATP synthase map in conformation-1 at pH 6

EMDB-28835: 
Yeast ATP synthase map in conformation-2, at pH 6

PDB-8f29: 
Yeast ATP synthase in conformation-1 at pH 6

PDB-8f39: 
Yeast ATP synthase in conformation-2, at pH 6

EMDB-29250: 
Yeast ATP Synthase in conformation-3, at pH 6

PDB-8fkj: 
Yeast ATP Synthase in conformation-3, at pH 6

EMDB-28685: 
Yeast ATP synthase consensus map in conformation-0 at pH 6

EMDB-29270: 
Yeast ATP Synthase map in presence of MgATP

EMDB-29278: 
F1-focused map of yeast ATP Synthase in presence of MgATP

PDB-8fl8: 
Yeast ATP Synthase structure in presence of MgATP

EMDB-29251: 
Fo and central rotor focused map of yeast ATP Synthase in conformation-3, at pH 6

EMDB-42455: 
Candidatus Methanomethylophilus alvus tRNAPyl in A-site of ribosome

PDB-8upt: 
Candidatus Methanomethylophilus alvus tRNAPyl in A-site of ribosome

EMDB-40477: 
KLHDC2 in complex with EloB and EloC

PDB-8sh2: 
KLHDC2 in complex with EloB and EloC

EMDB-28913: 
SARS-CoV-2 spike glycoprotein trimer bound with two nanobodies

EMDB-28914: 
SARS-CoV-2 spike glycoprotein trimer bound with two nanobodies (locally-refined)

EMDB-28930: 
SARS-CoV-2 spike glycoprotein trimer with nanobody bound to RBD in "up" conformation

EMDB-28931: 
SARS-CoV-2 spike glycoprotein trimer with nanobody bound to RBD in the "up" conformation (locally-refined)

EMDB-28811: 
F1-focused map of yeast ATP synthase in conformation-1, at pH 6

EMDB-28814: 
Fo and central rotor focused map of yeast ATP synthase in conformation-1, at pH 6

EMDB-28836: 
F1-focused map of yeast ATP synthase in conformation-2, at pH 6

EMDB-28837: 
Fo and central rotor focused map of yeast ATP synthase in conformation-2, at pH 6

EMDB-28813: 
Fo-focused map of yeast ATP synthase in conformation-1, at pH 6

EMDB-28868: 
Overall, original map of SARS-CoV-2 spike glycoprotein trimer with RBDs in the down conformation with nanobody attached to two RBDs.

EMDB-28880: 
Locally refined map of SARS-CoV-2 spike glycoprotein trimer with nanobody bound to two RBDs

EMDB-28687: 
Yeast ATP synthase F1-focused map in conformation-0 at pH 6

EMDB-28689: 
Yeast ATP synthase Fo-focused map in conformation-0 at pH 6

EMDB-41097: 
cryoEM structure of Smc5/6 5mer
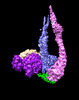
EMDB-41098: 
Smc5/6 8mer

PDB-8t8e: 
cryoEM structure of Smc5/6 5mer

PDB-8t8f: 
Smc5/6 8mer

EMDB-28856: 
SARS-CoV-2 spike protein trimer (down conformation) bound with a nanobody

PDB-8f4p: 
SARS-CoV-2 spike protein trimer (down conformation) bound with a nanobody

EMDB-29281: 
Cryo-EM structure of STING oligomer bound to cGAMP and NVS-STG2

EMDB-29282: 
Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53

PDB-8flk: 
Cryo-EM structure of STING oligomer bound to cGAMP and NVS-STG2

PDB-8flm: 
Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53

EMDB-28688: 
SARS-CoV-2 spike protein bound with a nanobody

PDB-8eyh: 
SARS-CoV-2 spike protein bound with a nanobody

EMDB-28686: 
SARS-CoV-2 spike protein complexed with two nanobodies

PDB-8eyg: 
SARS-CoV-2 spike protein complexed with two nanobodies

EMDB-15662: 
Cryo-EM structure of yeast mitochondrial RNA polymerase transcription initiation complex with 4-mer RNA, pppGpGpUpA (IC4)

EMDB-15664: 
Cryo-EM structure of yeast mitochondrial RNA polymerase transcription initiation complex with 5-mer RNA, pppGpGpApApA (IC5)

EMDB-15665: 
Cryo-EM structure of yeast mitochondrial RNA polymerase transcription initiation complex with 6-mer RNA, pppGpGpApApApU (IC6)

EMDB-16442: 
Cryo-EM structure of yeast mitochondrial RNA polymerase transcription initiation complex with 7-mer RNA, pppGpGpUpApApApU (IC7)
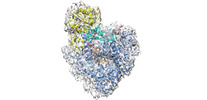
EMDB-16443: 
Cryo-EM structure of yeast mitochondrial RNA polymerase transcription initiation complex with 8-mer RNA, pppGpGpUpApApApUpG (IC8)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
